Статья размещена в открытом доступе и распространяется на условиях лицензии Creative Commons Attribution (CC BY).
МЕТОД
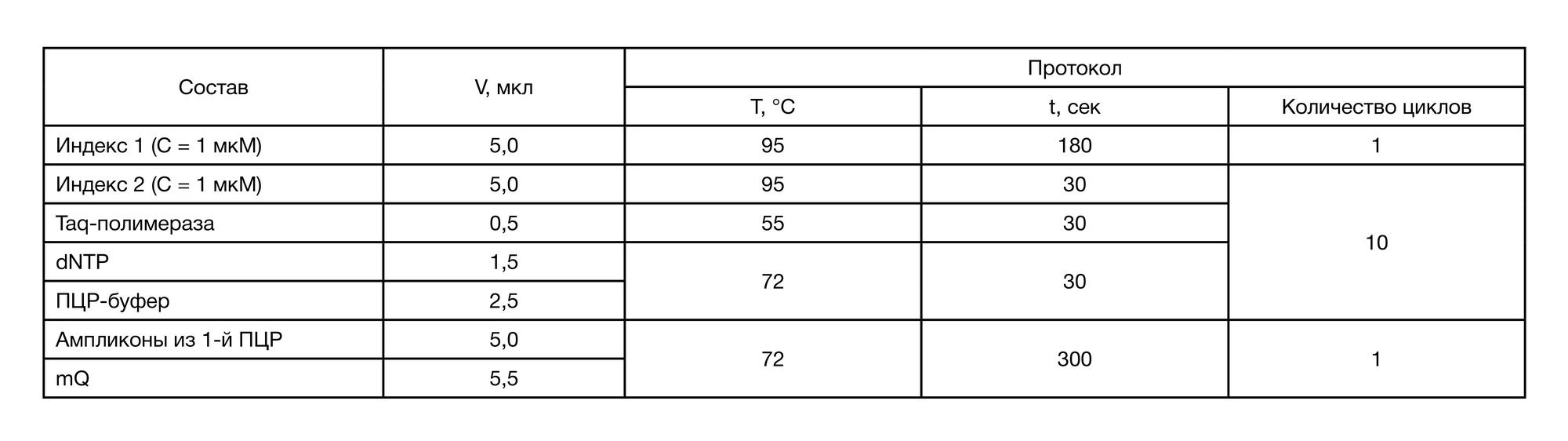
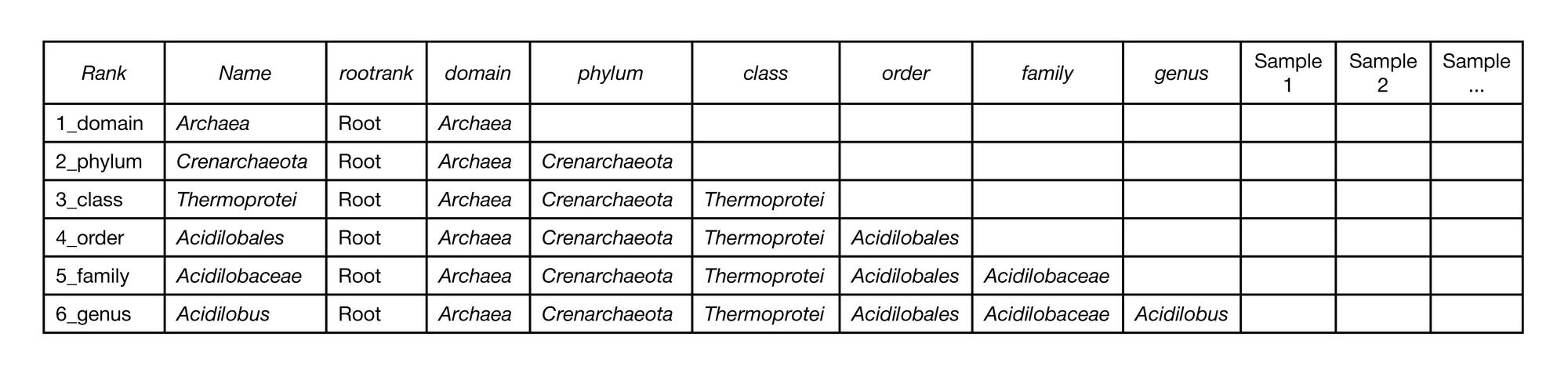
Особенности подготовки библиотек для метагеномного секвенирования образцов на платформе Illumina
1 ООО «Генотек», Москва
2 Московская гимназия на Юго-западе № 1543, Москва
Для корреспонденции: Красненко Анна Юрьевна
Наставнический пер., 17, стр. 1, подъезд 14, г. Москва, 105120; moc.liamg@oknensarkanna
Благодарности: авторы благодарят Дарью Плахину и Ивана Стеценка из ООО «Генотек» за помощь в работе, а также Сергея Глаголева из Московской гимназии на Юго-Западе № 1543 — за ценные советы и замечания.
Вклад авторов в работу: А. Ю. Красненко, А. Ю. Елисеев — анализ литературы, планирование и выполнение исследования, анализ и интерпретация данных; Д. И. Борисевич, К. Ю. Цуканов — биоинформатический анализ данных; А. И. Давыдова — подготовка черновика рукописи; В. В. Ильинский — планирование исследования, научный руководитель. Все авторы принимали участие во внесении исправлений в текст рукописи.