Статья размещена в открытом доступе и распространяется на условиях лицензии Creative Commons Attribution (CC BY).
ОРИГИНАЛЬНОЕ ИССЛЕДОВАНИЕ
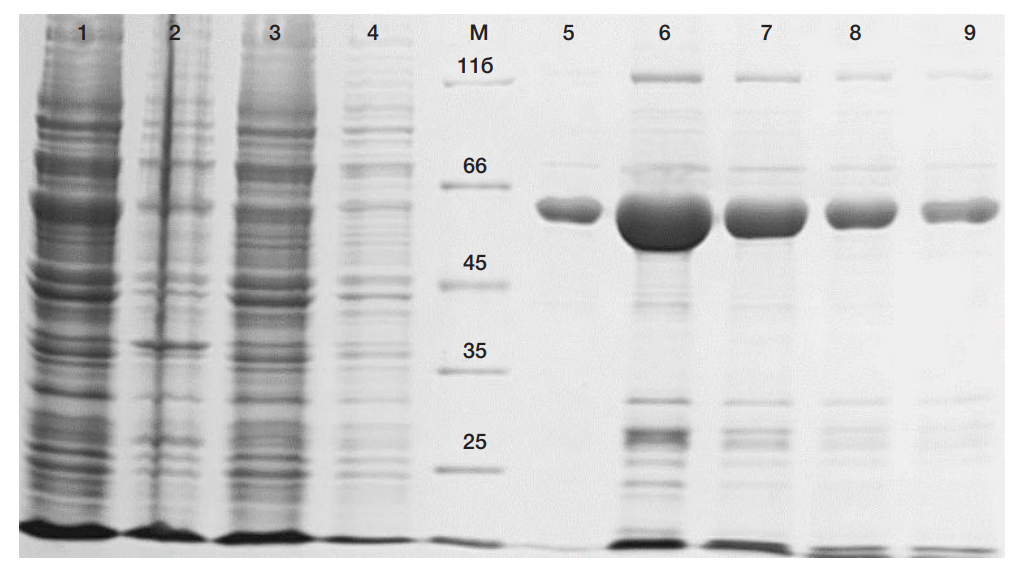
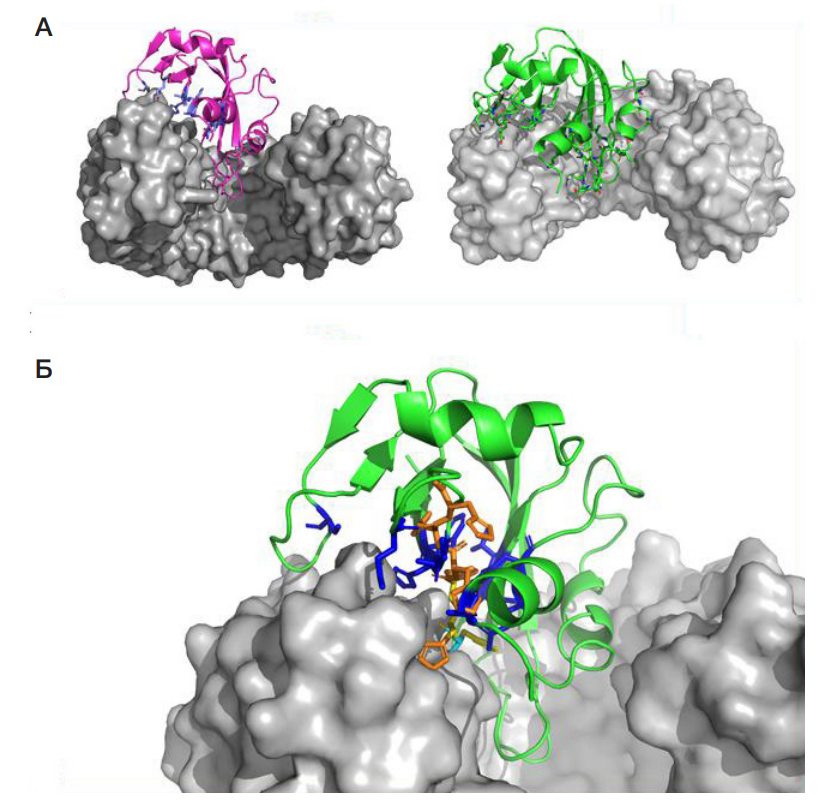
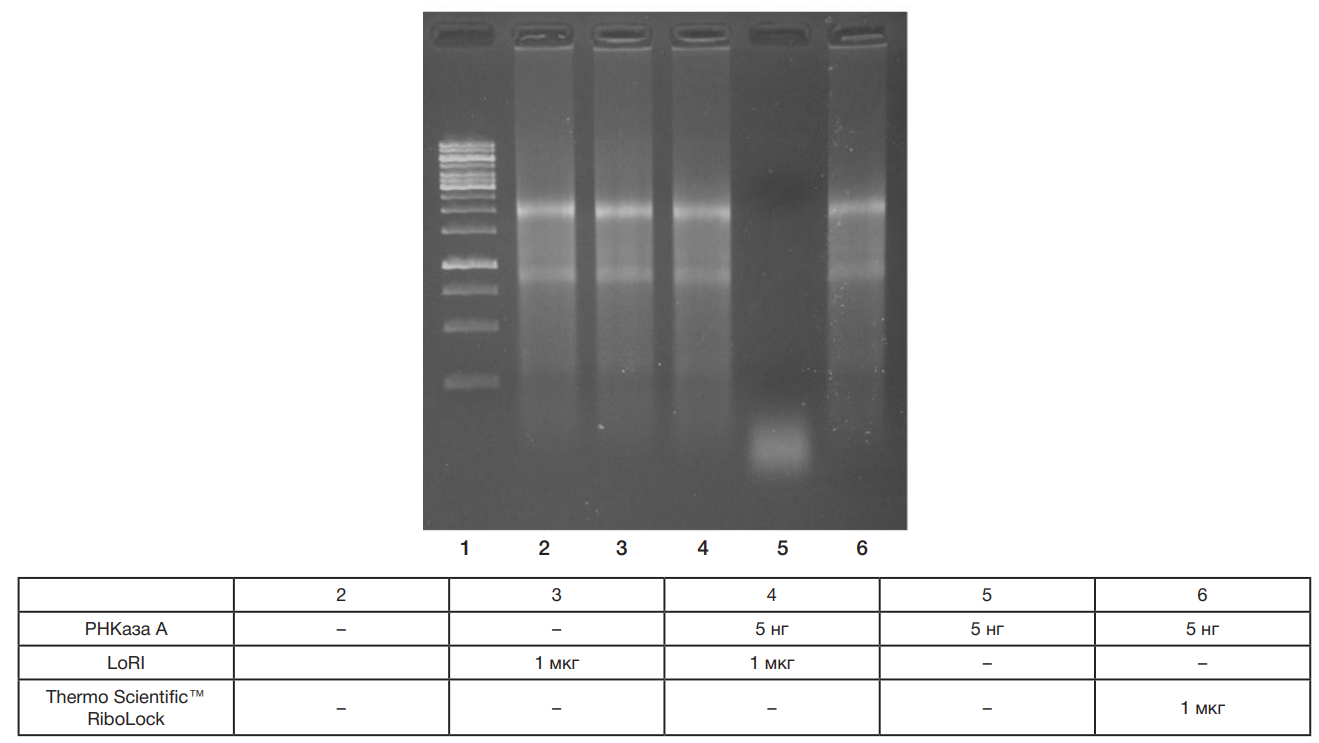
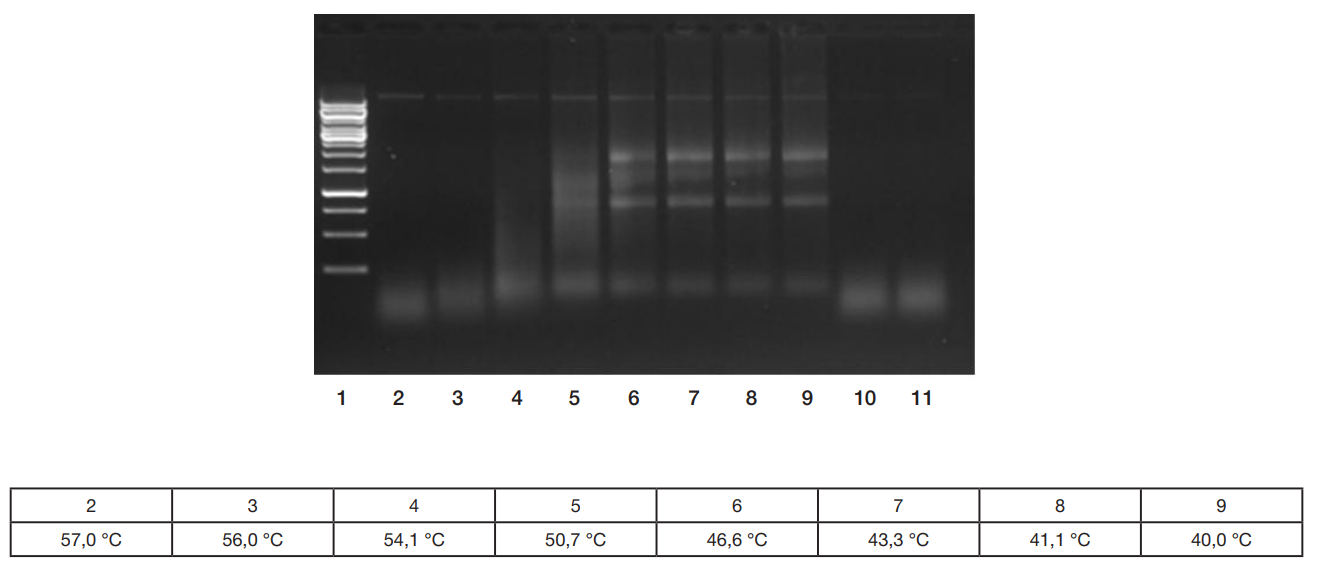
Новый рекомбинантный ингибитор РНКаз LoRI для применения in vitro
1 ООО «ДНК-Технология», Москва, Россия
2 МИРЭА — Российский технологический университет, Москва, Россия
3 Институт биоорганической химии имени М. М. Шемякина и Ю. А. Овчинникова, Москва, Россия
4 Московский государственный технический университет имени Н. Э. Баумана, Москва, Россия
5 Российский национальный исследовательский медицинский университет имени Н. И. Пирогова, Москва, Россия
Для корреспонденции: Наталья Юрьевна Усман
ул. Островитянова, д. 1/1, г. Москва, 117997, Россия; ur.relbmar@namsu_n
Финансирование: исследование выполнено при финансовой поддержке ООО «ДНК-Технология».
Вклад авторов: Д. А. Сухов, Г. А. Романенко — инженерия, экспрессия и очистка белков; И. В. Холошенко, Т. В. Петрова — характеризация продукта, написание статьи; М. Ю. Мышкин — анализ структуры белка; В. Ю. Кост — дизайн исследования; Д. Ю. Трофимов — руководство проектом; Н. Ю. Усман — написание статьи; Е. В. Барсова — координация проекта, написание статьи.